NCBI批量下载基因序列
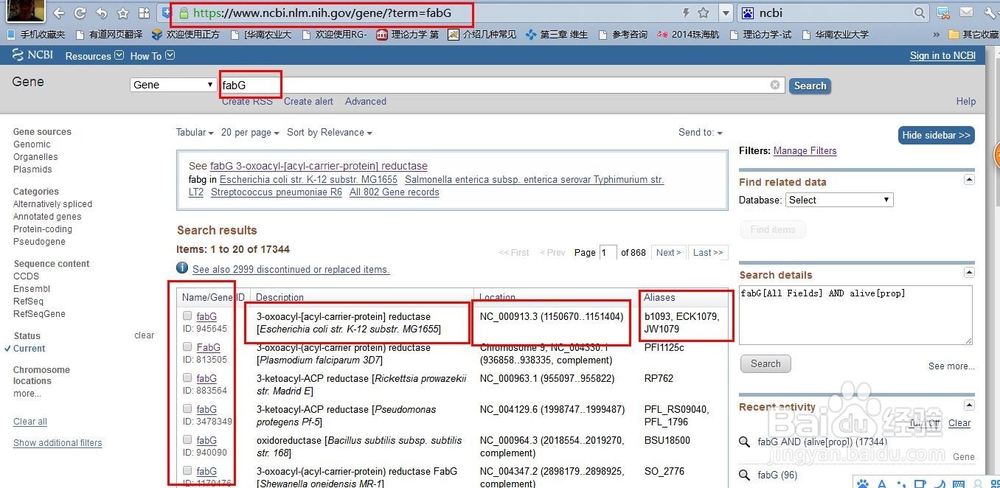
来源:网络收集 点击: 时间:2024-02-24我们需要通过这一下三步到达页面,获取信息。在Gene数据库中搜索fabG,然后进入基因界面,再点击基因进入序列界面。这三部构成了方法的基础。

 2/6
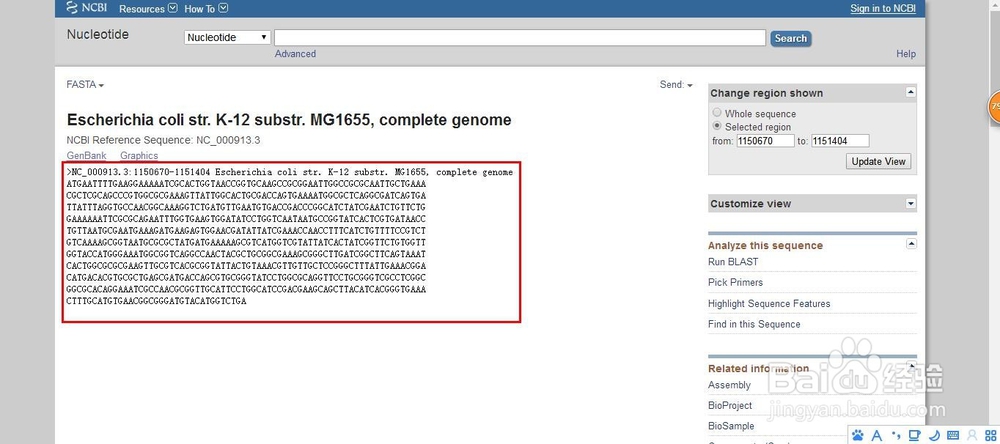
2/6通过源码分析,我们可以知道,先获取基因页面链接,再获取fasta页面链接,之后再获取序列。

 3/6
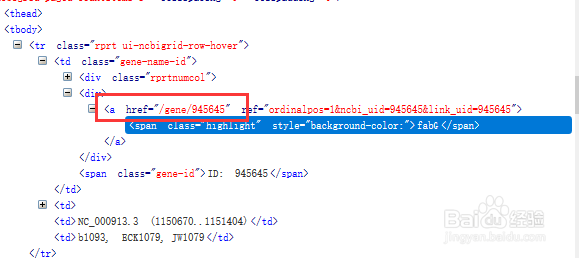
3/6构造此方法用于获取基因的链接列表。
从而可以进入基因的信息页面。
import urllib
import urllib.request
from urllib.error import HTTPError
from bs4 import BeautifulSoup
import re
import time
from selenium import webdriver
file=open(C:\\Users\\jyjh\\Desktop\\fabG\\allfabG.txt,w)
url=https://www.ncbi.nlm.nih.gov/gene/?term=fabG
geneurl=https://www.ncbi.nlm.nih.gov/
data=
html=urllib.request.urlopen(url)
bsObj=BeautifulSoup(html)
geneurllist=
for i in bsObj.find(tbody).findAll(tr):
geneurllist.append(geneurl+i.find(a))
 4/6
4/6for i in geneurllist:
html1=urllib.request.urlopen(i)
bsObj1=BeautifulSoup(html1)
print(bsObj1.findAll(h1).get_text().replace(,).strip())
temp=bsObj1.findAll(a)
fastaurl=
for i in temp:
if i.get_text()==FASTA:
if report in i:
fastaurl=i
break
driver=webdriver.PhantomJS(executable_path=C:\\Users\\jyjh\\Desktop\\phantomjs-2.1.1-windows\\bin\\phantomjs)
driver.get(geneurl+fastaurl)
time.sleep(3)
data.append(driver.find_element_by_id(viewercontent1).text)
driver.close()
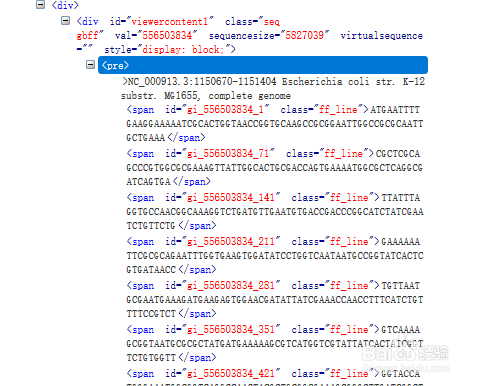
此方法用用于进入基因序列页面并获取数据
 5/6
5/6for i in data:
file.write(i+\n)
file.close()
写入数据到txt 文档中。并保存关闭。
 6/6
6/6其中最后的基因序列,只有在浏览器中才能看到,因为使用js和jq的方式生成的数据,在源代码中是不出现的。
运行结果如下图:

 注意事项
注意事项可以尝试不同的搜索
NCBI批量下载基因序列版权声明:
1、本文系转载,版权归原作者所有,旨在传递信息,不代表看本站的观点和立场。
2、本站仅提供信息发布平台,不承担相关法律责任。
3、若侵犯您的版权或隐私,请联系本站管理员删除。
4、文章链接:http://www.1haoku.cn/art_143390.html
 订阅
订阅