如何做pcoa图
来源:网络收集 点击: 时间:2024-04-06PCoA的作图主要分为三个步骤:选择特定的相似性距离并计算距离矩阵。距离的选择可以有Bray-curits、Unifrac等,不同的距离有不同的作用和意义(具体可以参考 微生物β多样性常用计算方法比较)。
 2/5
2/5相似性距离可以利用R的GUniFrac和vegan等包计算,也可以利用QIIME计算。
进行PCoA分析,也就是利用表征分析选择最能表示样本距离的坐标轴。这个可以利用R的ape包的pcoa()命令完成。
 3/5
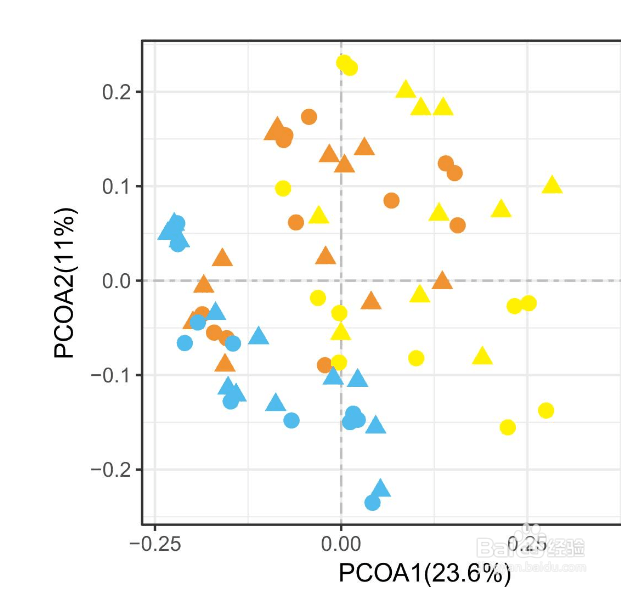
3/5PCoA图形展示。图形可以用ordiplot()命令展示,但如果需要比较美观的图形,建议用ggplot来画。
 4/5
4/5###导入需要的R包
library(GUniFrac)#用于计算Unifrac距离
library(ape) # 用于pcoa分析
library(ggplot2) #用于画图
##读文件
Otu_tab - read.table(otu_table,row.names=
Tree - read.tree(muscle.align.tree.nhx)=#输入OTU表格
Otu_tab -
Otu_tab_rff -
unifracs - gunifrac(otu_tab_rff,tree,alpha=c(0, 0.5,=
du - unifracs$unifracs=# 计算Unweighted UniFrac距离
 5/5
5/5Group - c(a,b,c)=#按照目的输入样本
shape - c(a=16,B =17,c=16)#定义点形状
color - c(a=#CCFF33 ,b=#CCFF33 ,c=#CCFF33 )=#定义点颜色
PCOA - pcoa(du,= correction=none ,= rn=NULL)#利用PCOA()指令做pcoa分析
 注意事项
注意事项网络虽好,但要注意劳逸结合哦!
如果是青少年,小编在这里提示大家千万不能沉迷网络!
版权声明:
1、本文系转载,版权归原作者所有,旨在传递信息,不代表看本站的观点和立场。
2、本站仅提供信息发布平台,不承担相关法律责任。
3、若侵犯您的版权或隐私,请联系本站管理员删除。
4、文章链接:http://www.1haoku.cn/art_446367.html
 订阅
订阅