科研/NCBI/根据蛋白序列查找某菌种同源基因信息
来源:网络收集 点击: 时间:2024-04-01首先登陆https://www.ncbi.nlm.nih.gov/pubmed,NCBI首页。点击首页下端BLAST,进入blast界面。
 2/7
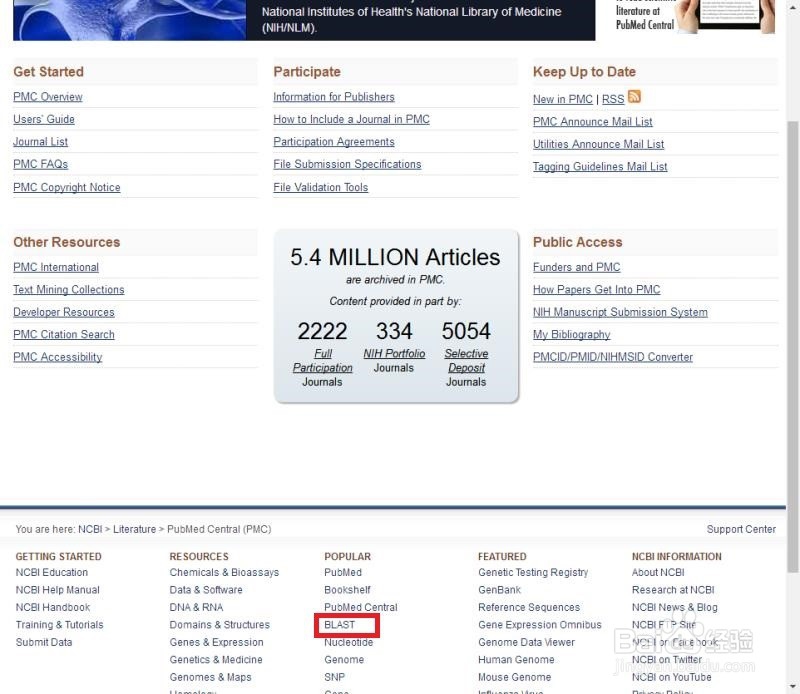
2/7BLAST界面提供blastn,blastp,blastx,tblastn,tblastx等选项。已知蛋白序列,查找某菌种对应的同源基因信息,点击tblastn
 3/7
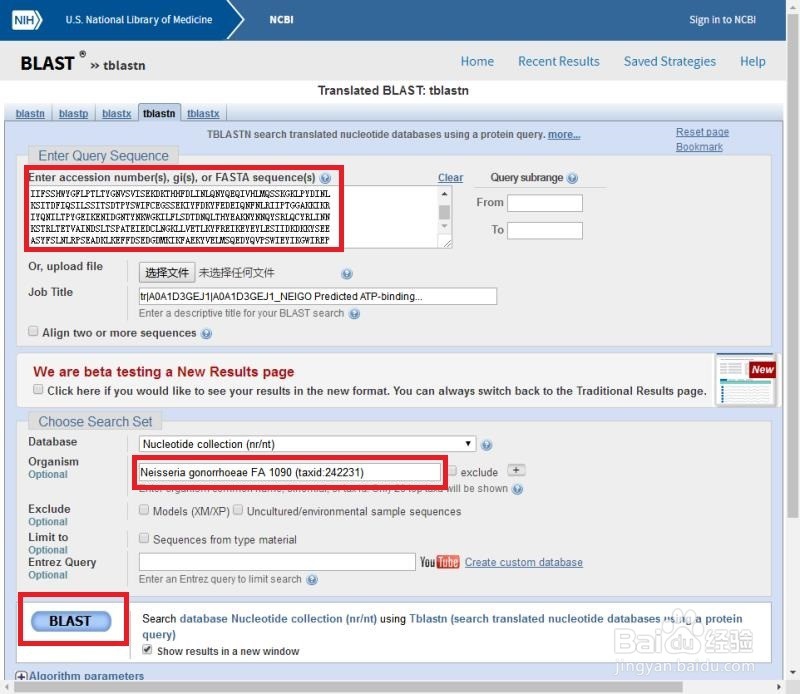
3/7点击进入后,复制蛋白序列到序列框,或者“选择文件”。选择Organism。完成上述信息后,点击blast。若有多个蛋白序列需要blast,建议勾选blast按钮旁边的“新窗口显示Show results in a new window”
 4/7
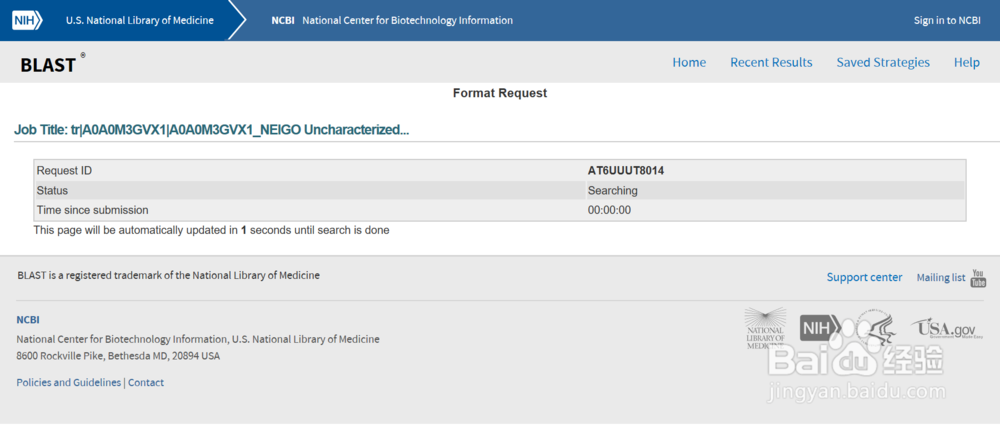
4/7跳转新页面后,网页会自动blast。每隔1s,2s,3s,7s等不停刷新网页,直至比对出结果,此时耐心等待。
 5/7
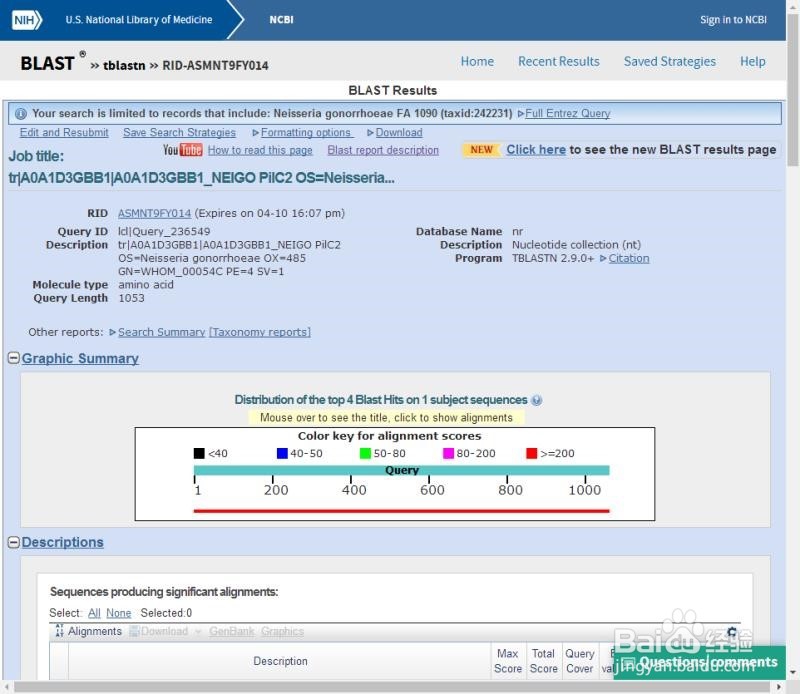
5/7比对结果完成后,页面显示如下。此时可查看结果。Sequences producing significant alignments栏中的Per. Inden越高越好。另一方面,Alignments栏下的Identities,Positives较重要。一般range1时比对结果最好的选择。此时可点击GeneBank跳转到genebank界面,查看对应基因信息。
 6/7
6/7有时genebank显示的时complete genome,此时拉到FEATURES查看香关具体信息。
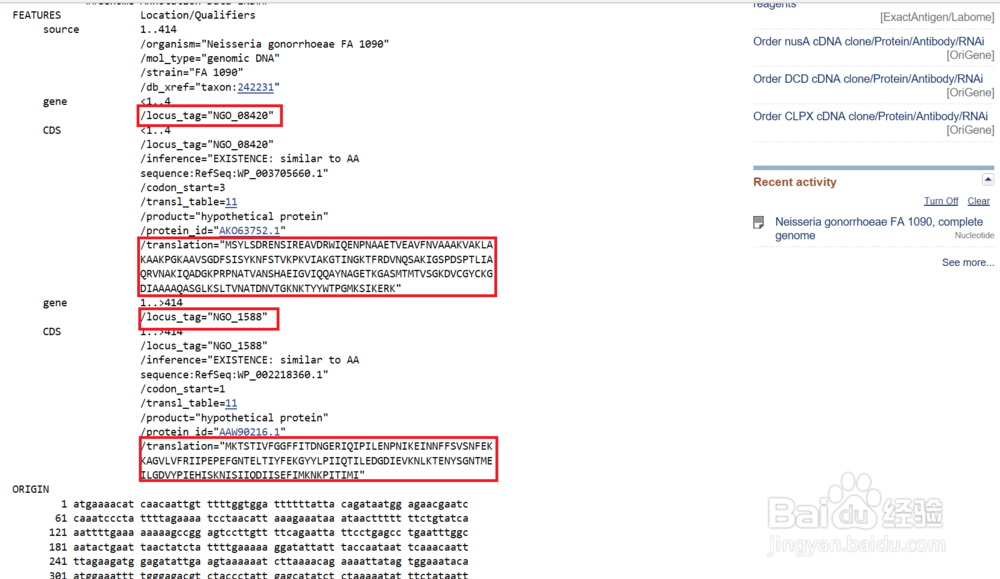 7/7
7/7ftp://ftp.ncbi.nlm.nih.gov/pub/factsheets/HowTo_NewBLAST.pdf
版权声明:
1、本文系转载,版权归原作者所有,旨在传递信息,不代表看本站的观点和立场。
2、本站仅提供信息发布平台,不承担相关法律责任。
3、若侵犯您的版权或隐私,请联系本站管理员删除。
4、文章链接:http://www.1haoku.cn/art_392063.html
 订阅
订阅